EUOS25 Challenge参加体験記(B01 大上班)

図1: 参加チームの集合写真
EU-OPENSCREENとSLASが主催する化合物AI予測コンペティション「EUOS25 Challenge」[1]において、大上班の学生で構成されたチームyumizがTransmittanceカテゴリーのブラインドテストで1位となりました(Fluorescenceカテゴリーでは6位でした)。
優勝した我々のチームには、ボストンで開催されるSLAS2026 International Conference & Exhibitionにて表彰され、副賞€1,000が授与されました。
また、優勝チームと、性能に有意差のない結果を出したチームは、SLAS journalにて研究またはプロトコルを発表するよう招待されています。
以降、我々の解法を紹介し、コンペティションを通じて得られた経験と知見を共有したいと思います。
なお、実際のコード等はohuelab/euos25-solutionにて公開しているので詳細はそちらを参照してください。
コンペ概要
EUOS25 Challengeは化合物の分光特性、透過率(吸収)および蛍光を予測するデータサイエンスコンペティションです。
数万件の訓練データを用いて、Transmittanceタスクは特定波長における光の吸収の有無を、Fluorescenceタスクは蛍光の有無を予測する二値分類タスクとなっています。TransmittanceとFluorescenceの2つのカテゴリーでそれぞれ順位が決定されました。
それぞれ以下のように2つのサブタスクに分かれており、各サブタスクのROC AUCの平均で順位が決まります。
- Transmittanceサブタスク1a: 340 nmで透過率≤70%の化合物を予測
- Transmittanceサブタスク1b: 450~679 nmの範囲で平均して透過率≤70%の化合物を予測
- Fluorescenceサブタスク2a: 励起340 nm・発光450 nmの条件で、蛍光強度がしきい値を超える化合物を予測
- Fluorescenceサブタスク2b: 励起/発光の波長ペアが480/540、525/598、560/610 nmのいずれかの条件で、蛍光強度がしきい値を超える化合物を予測
特に、サブタスク1bと2bでは深刻なラベル不均衡が存在することが知られており(透過率タスクでは約1.5%、蛍光タスクでは約0.23%のみが正例)、これらのサブタスクにおいても高い性能を発揮するモデルをいかに構築できるかが本コンペティションの主要な課題の一つでした。
コンペティションは2025年10月から2026年1月まで開催されました。
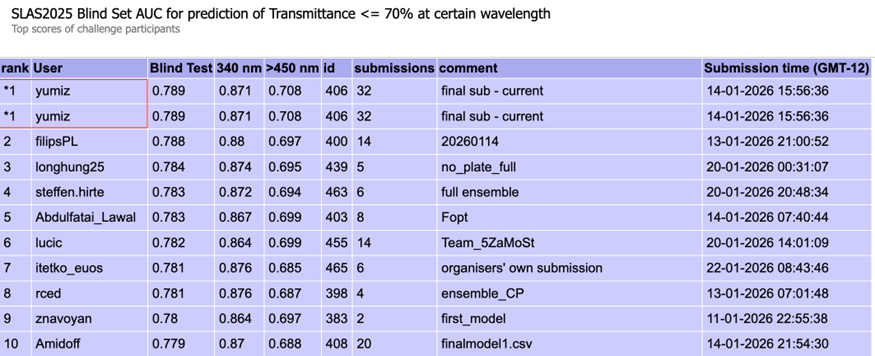
図2: Transmittanceタスクのブラインドテスト結果
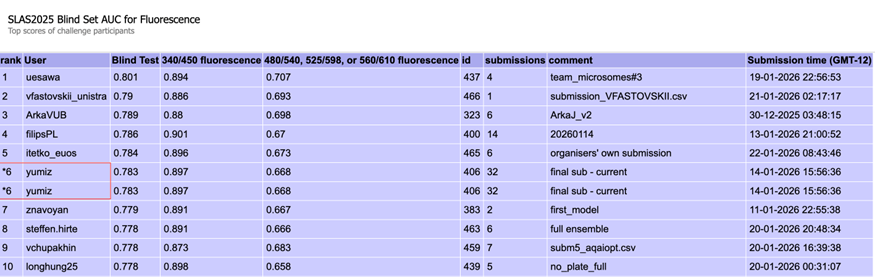
図3: Fluorescenceタスクのブラインドテスト結果
解法
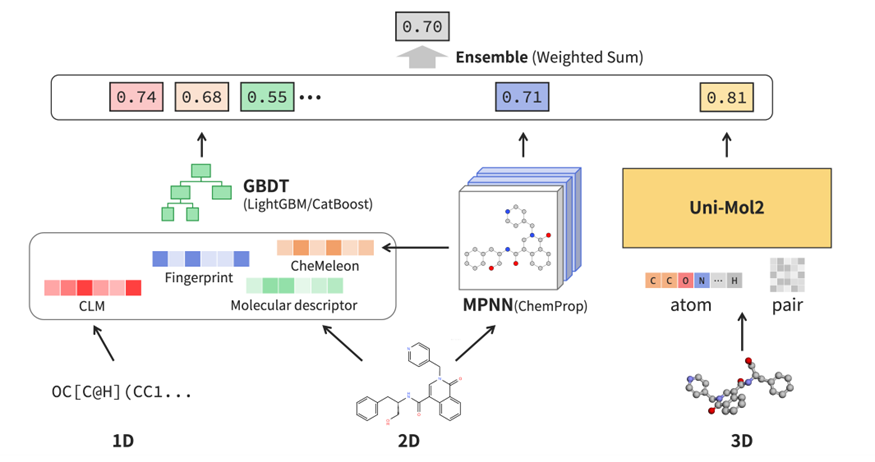
図4: 大上研究室の優勝解法の概要図
大上研究室では、1次元・2次元・3次元の分子情報を統合したマルチモーダル分子特性予測と、複数のモデルアーキテクチャを用いた重み付けアンサンブルを実装しました。
まず、LightGBMやCatBoostなどの勾配ブースティング決定木(GBDT)を用いた予測モデルを、以下の特徴量を単独または様々に組み合わせたパターンで学習し、交差検証性能が高いモデルを採用しました。
- 化合物言語モデル(ChemBERTa[2])の潜在表現
- RDKitやMordredで計算した化合物記述子
- ECFP4を用いた分子フィンガープリント
- CheMeleon[3](Mordred記述子で事前学習したChemPropベースのMPNNモデル)の潜在表現
さらに、ChemProp[4]やCheMeleonを用いた予測モデルも構築しました。また、Uni-Mol2[5](84M, 310M)を用いて、3次元構造を利用した予測モデルも構築しました。
複数のモデルからの予測を効率的に統合するため、交差検証スコアに基づいて重み付け和によるアンサンブルを行い、精度の高い組み合わせを選択して提出しました。
このように、分子の1次元情報(SMILES)、2次元情報(構造)、3次元情報(3Dコンフォーメーション)を含むマルチモーダルな分子表現を利用しています。
それから、コアモデルに加えて、以下のような前処理、学習、最適化手法も適用しました。
- 塩の除去などの基本的なSMILES前処理
- 透過率タスクにおける回帰モデルの使用を検討
- Focal Lossを用いた不均衡データへの対処
- ChemPropを用いたマルチタスク学習
- Murcko scaffoldsデータ分割による5分割交差検証
- GBDTモデルに対するnested cross validation + Optunaによるハイパーパラメータチューニング
特にMurcko scaffoldsデータ分割による交差検証は、モデルの汎化性能を適切に評価するのに重要でした。
分子の基本骨格(scaffold)が似ている化合物を同じデータセット(訓練/検証)に含めることで、データリークを防ぎ、訓練データにないscaffoldを持つ化合物に対する予測性能を適切に評価できます。
結果
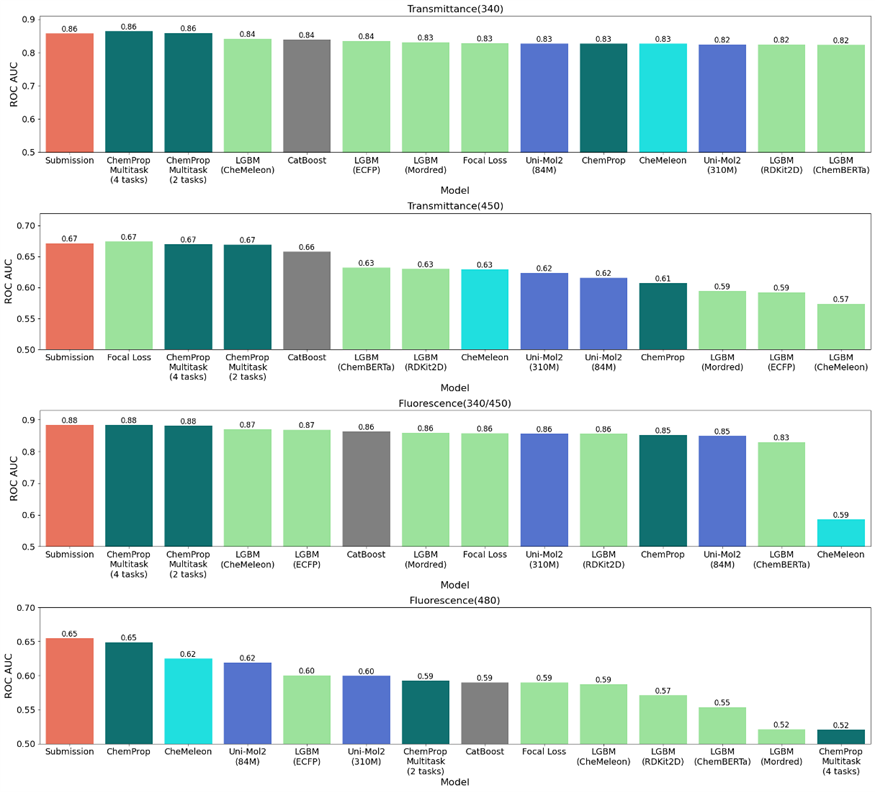
図5: 各タスクにおけるの手法ごとのリーダーボードでの性能
図5はコンペティション終了後にリーダーボードのテストセットを用いて、複数の単独モデルの予測性能と最終提出モデルの予測性能を比較したものです。
全てのタスクにおいて一貫した傾向は見られませんでしたが、最終提出モデルの性能は一貫して良好でした。
多様な情報源をアンサンブルによって組み合わせることで、頑健で汎化性能の高い予測モデルの構築に繋がったと考えられます。
また、表1にパブリックおよびプライベートリーダーボードでのスコアを示します。
パブリックリーダーボードとはコンペティション中に公開されるテストセットの一部で評価されたスコアで、プライベートリーダーボードはコンペティション終了後に公開されるテストセットの残りで評価されたスコアです。
全てのタスクにおいて、プライベートスコアはパブリックスコアを上回りました。
パブリックリーダーボードで我々よりも上位にランクインしていた多くのチームは、パブリックリーダーボードに過適合していた可能性が高いと考えられます。
パブリックからプライベートでの順位変動がこれほど大きかったことはやや予想外でしたが、scaffold分割によって適切に汎化性能を検証したことが優勝できた一因かもしれません。
表1: Public/Privateリーダーボードでのスコア
| タスク | Public | Private |
| Transmittance subtask 1a | 0.857 | 0.871 |
| Transmittance subtask 1b | 0.670 | 0.708 |
| Fluorescence subtask 2a | 0.883 | 0.897 |
| Fluorescence subtask 2b | 0.655 | 0.668 |
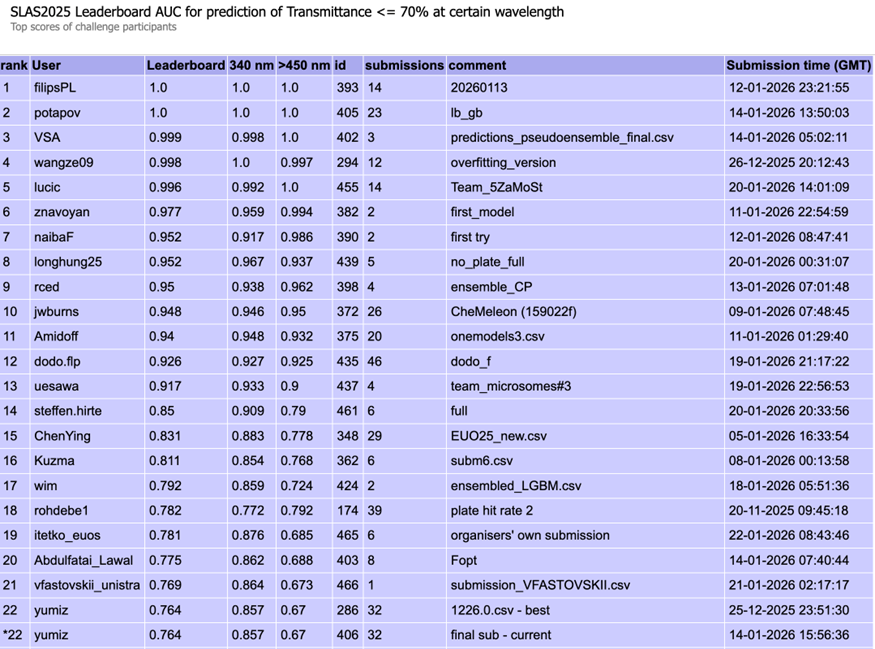
図6: Transmittanceタスクのパブリックリーダーボードの順位
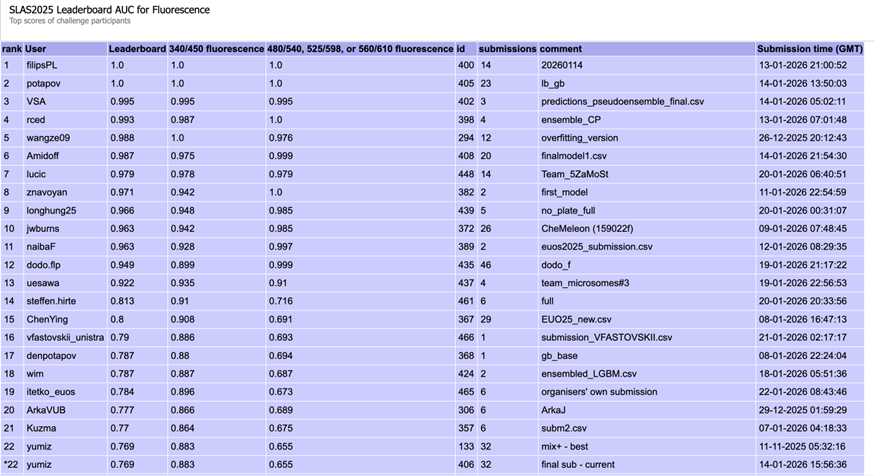
図7: Fluorescenceタスクのパブリックリーダーボードの順位
おわりに
以上のように、1次元・2次元・3次元の分子情報を統合したマルチモーダルな分子表現と、scaffold分割による適切な汎化性能の評価を組み合わせた重み付けアンサンブルアプローチが本コンペティションにおいて有用でした。
本コンペティションで得られた知見や経験は、本領域の研究においても役立つものと考えています。
このような有益なコンペティションを開催していただいた主催者の方々や、コンペティションを通じてアイデアや議論を共有してくれたチームメンバーに深く感謝申し上げます。
参考文献
[1] EUOS25 Challenge | Online Chemical Modeling Environment. https://ochem.eu/static/challenge2025.do
[2] Chithrananda, Seyone, et al. "ChemBERTa: Large-scale self-supervised pretraining for molecular property prediction." arXiv [cs.LG], 19 Oct. 2020. http://arxiv.org/abs/2010.09885
[3] Burns, Jackson, et al. "Descriptor-Based Foundation Models for Molecular Property Prediction." arXiv [Cs.LG], 18 June 2025. https://doi.org/10.48550/arXiv.2506.15792
[4] Heid, Esther, et al. "Chemprop: A Machine Learning Package for Chemical Property Prediction." Journal of Chemical Information and Modeling, vol. 64, no. 1, Jan. 2024, pp. 9–17. https://doi.org/10.1021/acs.jcim.3c01250
[5] Ji, Xiaohong, et al. "Exploring Molecular Pretraining Model at Scale." The Thirty-Eighth Annual Conference on Neural Information Processing Systems, 2024. https://openreview.net/pdf?id=64V40K2fDv

